Dies ist/wird eine Anleitung für Eveline, wie die Alveolen in den Acini zu zählen sind.
Alveolen zählen
Die Alveolen werden mit dem STEPanizer gezählt. Um die Alveolen zu zählen, muss die Beta-Version mit aktiviertem Disector verwendet werden (unterer “Start”-Link).
Proben-"Standorte"
- Probe 60A wurde nicht gemessen
Bilder exportieren mit DICOMReader
<note>Wurde schon gemacht</note> <note tip>
- Acini sind auf R gespeichert
- wurden mit MATLAB-Skript DICOMReader exportiert.
- in jedem Verzeichnis liegen die Daten zum auszählen in Unter-Ordnern namens “acinus01”, etc.
</note>
Die DICOM-Bilder, die aus MeVisLab exportiert wurden, werden mit dem Skript in JPG-Bilderstapel exportiert, die mit dem STEPanizer geladen werden können. Das Skript (DICOMReader) exportiert (bei Option Disector=1 auf Zeile 20) die Slices aus den Originaldaten mit definierbarer Disector-Dicke und Slice-Abstand.
 Die Erklärung der Parameter DisectorThickness und SliceDistance (auf Zeilen 21 und 22) ist im Bild rechts zu sehen (Oder in diesem pdf hier, das mit diesem LaTeX-File erstellt wurde).
Die Erklärung der Parameter DisectorThickness und SliceDistance (auf Zeilen 21 und 22) ist im Bild rechts zu sehen (Oder in diesem pdf hier, das mit diesem LaTeX-File erstellt wurde).
Das Skript exportiert für jeden Aufruf alle einzelnen Acini in Unterordner im Originalverzeichnis der Probe, die im Ordnernamen die Details der Parameter enthalten, also z.B.
...\R108C60B_B1_mrg\acinus01\voxelsize1.48-every10slice-DisectorThickness-4.44um-or3slices\ ...\R108C60B_B1_mrg\acinus01\voxelsize1.48-every6slice-DisectorThickness-7.40um-or5slices\ ...\R108C60B_B1_mrg\acinus01\voxelsize1.48-every10slice-DisectorThickness-4.44um-or3slices\
je nachdem, was für Parameter beim Aufruf eingegeben wurden.
Im Gespräch mit Stefan hat sich gezeigt, dass eine DisectorThickness von 3 Slices und eine SliceDistance von 10 Slices gut ist. Ich habe alle extrahierten Acini der Tiere von Tag 60 mit diesen Parametern auf das Laufwerk R geschrieben. Die einzelnen Links sind oben bei Proben-“Standorte” ersichtlich. In diesen Verzeichnissen hat's jeweils pro Acinus einen Unterordner “acinusX”, der nebst dem Verzeichnis voxelsize1.48-every6slice, welches für die Volumenbestimmung der Acini verwendet wurde, ein Verzeicznis voxelsize1.48-every10slice-DisectorThickness-4.44um-or3slices, welches die Daten enthält, welche mit den oben beschriebenen Parametern gespeichert wurden.
STEPanizer
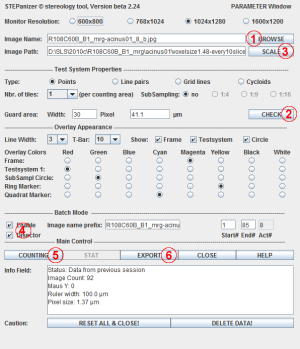 Im STEPanizer müssen die Bilder geladen werden, die Parameter eingestellt und überprüft werden, und anschliessend kann gezählt werden. Ich habe das immer ensprechend dem Bild rechts und dem Ablauf unten gemacht.
Im STEPanizer müssen die Bilder geladen werden, die Parameter eingestellt und überprüft werden, und anschliessend kann gezählt werden. Ich habe das immer ensprechend dem Bild rechts und dem Ablauf unten gemacht.
- Browse - Mit Browse wird der Ordner des Acinus ausgewählt, der gezählt werden soll. Am besten wird das erste Bild im gewünschten Ordner angeklickt.
- Check - Mit Check kann überprüft werden, ob die Einstellungen des Testsystems stimmen. Für die Zählung der Alveolen brauchen wir kein Zählsystem, das Check dient also nur dazu, das Bild erstmal korrekt zu laden
- Batch Mode Enable und Disector - Mit diesen beiden Feldern wird eingestellt, dass flink viele Bilder im Disector-Modus durchgezählt werden können. Am einfachsten wird der Dateinamen aus dem Image Name oben kopiert und hier eingefügt. Dann zuhinterst alles löschen bis zum zweitletzten “_”. In dem im Bild angegebenen Fall würde im Image name prefix: “R108C60B_B1_mrg-acinus01_” eingegeben. Im Start# und End#-Feld wird angegeben, welche Bilder ausgezählt werden. Beim im Bild angegebenen Fall (Probe 60B, Acinus 1) von Bild 1 bis 84 (oder 85, das ist nicht schlimm :)
- Counting - Mit Counting wird das zählen gestartet. Das haben wir zusammen angeschaut.
- Export - Mit Export werden nach dem zählen die Daten exportiert, siehe unten.
Auswertung
Nach dem auszählen mit dem STEPanzizer wird mit dem Export-Knopf eine .csv-Datei in das Verzeichnis des gerade gezählten Acinus geschrieben. Diese Datei kann einfach durch Doppelklick in Excel geöffnet werden, so dass die gezählten Brücken einfach in eine gesammelte Tabelle exportiert werden können.
Die Anzahl der Alveolen pro Acinus kann mit untenstehender Formel ausgerechnet werden.
Die Volumina der einzelnen Acini sind immer in den Dateinamen der DICOM-Bilder angegeben. So ist z.B. der erste Acinus der Probe 60B, der oben im STEPanizer-Fenster angegeben wurde, mit dem Filenamen
R:\SLS\2010c\R108C60B_B1_mrg\R108C60B_B1_mrg.2948.2948.1024.gvr.acinus1.volume0.30373824.pixelsize0.00148000009357929.dcm
auf der Festplatte gespeichert.
Das heisst,
- die Probe R108C60B wurde in der Beamtime 2010c als stacked Widefieldscan gemessen. B1 ist die oberste Schicht von drei Schichten des Stacks, mrg zeigt an, dass wir die Probe als WideFieldScan gemessen haben.
- die Originaldatei ist 2948x2948x1024 Pixel gross
- das mit MeVisLab bestimmtes Volumen des ersten Acinus ist 0.30373824 µl, bei einem Pixelsize von 0.00148 mm, was 1.48 µm entspricht. Die überzähligen Nachkommastellen kommen von interner Rundung und können vernachlässigt werden, die Pixelgrösse ist fix 1.48 µm.
Die Volumina habe ich in einer XLS-Tabelle zusammengetragen, die auch auf dem U: gespeichert (\\u:\Gruppe_Schittny\doc\David\AcinarVolumes.xls) und in meinem Laborbuch abgelegt ist. Dies Tabelle habe ich mit diesem MATLAB-File erstellt, welches die Filenamen ausliest und in einer Tabelle zusammenträgt, so muss das nicht mehr von Hand gemacht werden. Diese Tabelle enthält alle mit MeVisLab gemessenen Acinus-Volumina von Tag 60.
<note> Bei verschiedenen Acini habe ich das Volumen, welches mit MeVisLab automatisch anhand der Segmentierung bestimmt wurde, mit dem STEPanizer anhand der Cavalieri-Methode kontrolliert. Dieser Vergleich ist in diesem Excel-File zu finden (\\u:\Gruppe_Schittny\doc\David\AcinusGrössenVergleichSTEPanizer.xls). Bei einem evtl. gewünschten Vergleich der MeVis-Messung mit dem STEPanizer ist es wichtig zu schauen, von welchen Acini das Volumen bisher schon ausgezählt wurde, es wurden bei weitem nicht alle stereologisch vermessen! </note>
Mit der untenstehenden Formel wurde aus den Points, die innerhalb der segmentierten Acni gezählt wurden, das Volumen derselben bestimmt. Diese Daten wurden in einem XLS-File zusammengetragen, damit der vergleich zwischen MeVisLab und dem STEPanizer gemacht werden konnte. Dieses XLS-File ist noch auf meinem P-Laufwerk zu finden (AcinusGrössenVergleichSTEPanizer.xls), das es für diese Messung nicht relevant ist, habe ich es nicht aufs U-Laufwerk kopiert.
